采用doccano的目的是为了进行人工标记
采用Linux操作系统的Docker安装,用的84端口,账号:admin,密码:123,邮箱:qq
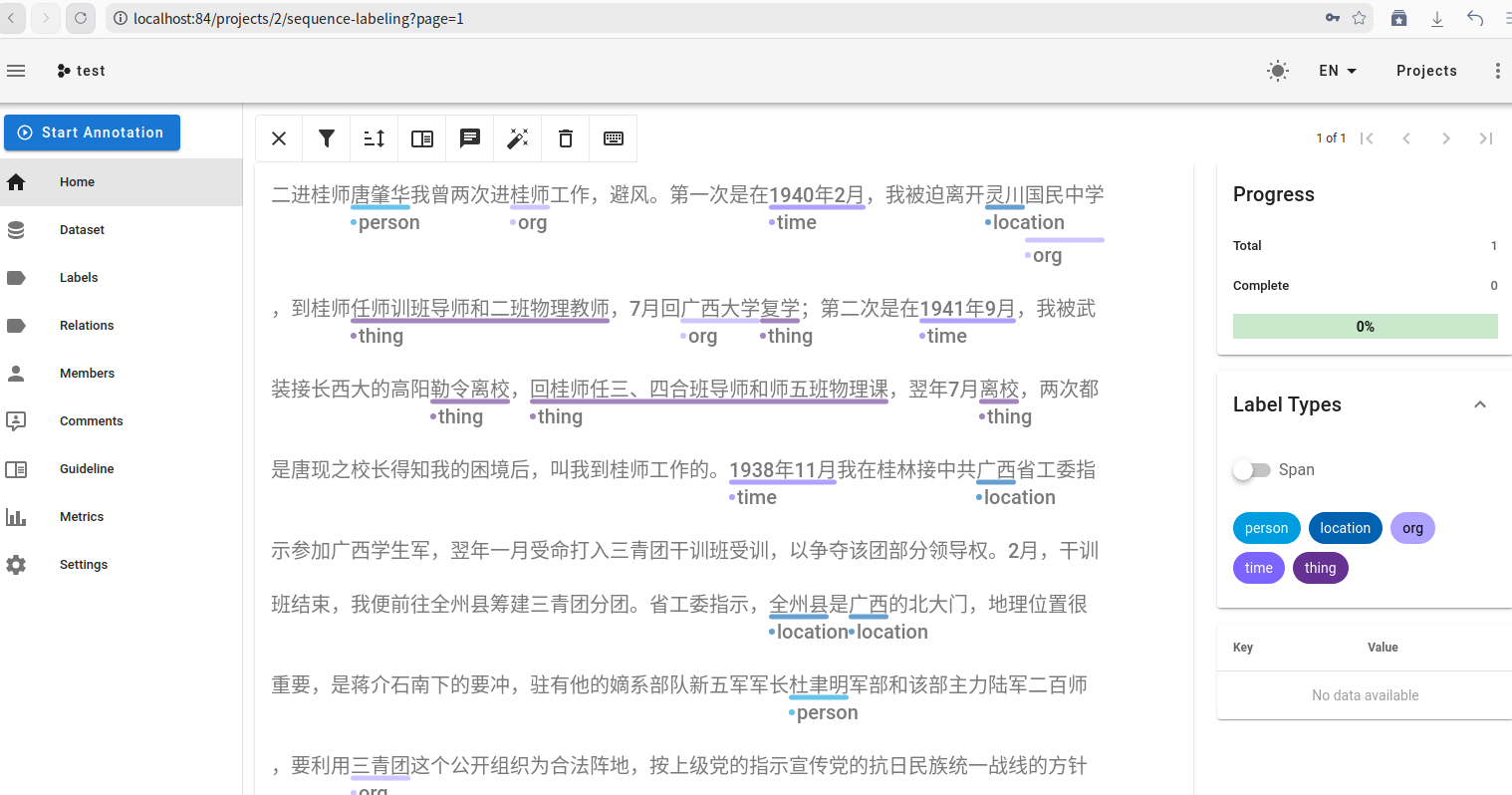
导入clean.txt文件 ,然后采用5个标志,做一些简要标注,如图
首先仅针对第一篇回忆文章进行分析 ,进行历史档案资源本体构建、知识图谱构建等工作
对一篇历史档案资源文档进行本体构建,需要系统地规划和执行,具体步骤包括:确定目标、本体设计、知识提取、关系建模、存储实现及应用开发。以下是详细的流程和所需技术:
(1)定义档案资源中的核心概念(如时间、事件、人物、地点)。
(2)揭示档案资源中概念之间的关系(如人物与事件的参与关系、事件与地点的关联)。
(3)为后续的知识检索、推理、关联分析提供结构化知识支持。
(1)明确要覆盖的档案内容:例如,特定历史时期、区域的事件记录。
(2)选择粒度:是否关注微观细节(如个体人物)或宏观结构(如历史时期的关键事件)。
对历史档案文档进行清洗和语义分析,提取知识所需的基本信息。
### 技术需求 -
OCR(光学字符识别):如果档案文档是扫描版或图像格式,使用OCR工具如Tesseract提取文本。
-
自然语言处理(NLP):使用工具进行文本分词、实体识别(NER)、关系抽取。
- 推荐工具: - HanLP:适合中文语料的分词和实体识别。 -
spaCy:可用于多语言实体识别。 -
OpenNRE:开源关系抽取工具。 -
知识图谱基础技术:从文档中提取三元组(subject-predicate-object)。
本体是领域知识的概念化表示,需定义概念及其关系。
从档案文本中提取实体和关系,生成三元组。
从文本中提取重要实体(如时间、地点、人物、事件)。 - 工具:HanLP、spaCy、jieba分词结合NER。
通过上下文语义或预训练模型识别实体间的关系。 - 示例:从“杜聿明指挥新五军参加桂南会战”中提取三元组: - (杜聿明, 指挥, 新五军) - (新五军, 参加, 桂南会战)
将本体数据存储为结构化格式,以便进行查询和推理。
将三元组数据导入存储工具中,构建初步知识库。
利用本体和知识库,实现推理、检索和可视化。
通过推理引擎,基于已定义的关系和规则生成新知识。 - 推理引擎: - Apache Jena:支持RDF/OWL推理。 - RDF4J:轻量级语义推理工具。 - SPARQL:用于查询知识图谱。
以下是完整工具链的推荐: | 类别 | 工具/框架 | 作用 | |——————–|———————————–|—————————–| | 文本处理 | HanLP、jieba、spaCy | 分词、实体识别 | | 知识提取 | OpenNRE、pykg2vec、BERT | 实体与关系抽取 | | 本体建模 | Protégé、OWL API、Neo4j | 本体设计与存储 | | 图数据库 | Neo4j、GraphDB、Amazon Neptune | 知识图谱存储与查询 | | 推理引擎 | Apache Jena、RDF4J | 语义推理 | | 可视化 | Gephi、D3.js、Cytoscape | 关系网络、知识图谱展示 |
针对第一篇已经处理有文字信息的历史文档,资源的核心概念是时间、事件、人物、地点,我想要提示人物与事件的参与关系、事件与地点的关联,应该具体如何去做本体构建。 步骤 1:对文档清洗和分段。 步骤 2:通过NER提取实体(人物、地点、时间、事件)。 步骤 3:通过上下文提取人物与事件、事件与地点的关系。 步骤 4:构建本体并存储为RDF文件。 步骤 5:通过图数据库或SPARQL查询实现可视化与语义检索。
针对目前通过NER提取实体(人物、地点、时间、事件)时,只能提取到人物、时间,而无法提取到事件、地点等信息,而进行优化解决。
Doccano 是一个开源、基于 Web 的标注工具,支持文本分类、序列标注(命名实体识别),以及文本关系标注任务。以下是从安装到使用的完整流程:
安装 Docker 和 Docker Compose。
创建一个
docker-compose.yml文件,内容如下:
version: "3"
services:
doccano:
image: doccano/doccano
ports:
- "8000:8000"
environment:
- ADMIN_USERNAME=admin
- ADMIN_PASSWORD=password
- ADMIN_EMAIL=admin@example.com启动服务:
docker-compose up -d访问 Doccano: 打开浏览器,访问
http://localhost:8000,用 admin 和
password 登录。
创建虚拟环境:
python3 -m venv doccano_env
source doccano_env/bin/activate安装 Doccano:
pip install doccano启动服务:
doccano init
doccano createuser --username admin --password password --email admin@example.com
doccano runserver打开浏览器访问 http://localhost:8000。
点击 “Upload Data”。
上传数据文件(支持 JSON、CSV、TXT 等格式)。
TXT 文件格式
:
毛泽东于1927年9月9日领导秋收起义。
后率军上井冈山。JSON 文件格式
:
[
{"text": "毛泽东于1927年9月9日领导秋收起义。"},
{"text": "后率军上井冈山。"}
]完成标注后,点击 “Download Data”。
选择导出格式(JSON、CSV 等)。
JSON 输出示例
:
[
{
"id": 1,
"text": "毛泽东于1927年9月9日领导秋收起义。",
"entities": [
{"start": 0, "end": 3, "label": "人物"},
{"start": 5, "end": 15, "label": "时间"},
{"start": 16, "end": 20, "label": "事件"}
]
}
]Doccano 提供了 RESTful API,可以通过代码实现数据导入、标注管理和导出。
安装
requests库:
pip install requests登录获取 Token:
import requests
url = "http://localhost:8000/api/v1/auth/login/"
data = {"username": "admin", "password": "password"}
response = requests.post(url, json=data)
token = response.json()["token"]创建项目:
headers = {"Authorization": f"Token {token}"}
project_url = "http://localhost:8000/api/v1/projects/"
project_data = {"name": "NER Project", "description": "Entity Recognition", "task_type": "SequenceLabeling"}
response = requests.post(project_url, json=project_data, headers=headers)
print(response.json())导入数据:
project_id = response.json()["id"]
upload_url = f"http://localhost:8000/api/v1/projects/{project_id}/docs/"
documents = [{"text": "毛泽东于1927年9月9日领导秋收起义。"}]
requests.post(upload_url, json=documents, headers=headers)导出标注结果:
export_url = f"http://localhost:8000/api/v1/projects/{project_id}/export?format=JSON"
response = requests.get(export_url, headers=headers)
print(response.json())采用doccano的目的是为了进行人工标记
采用Linux操作系统的Docker安装,用的84端口,账号:admin,密码:123,邮箱:qq
导入clean.txt文件 ,然后采用5个标志,做一些简要标注,如图

然后是进行机器学习模型训练或分析
采用BERT学习模型,用到BIO格式文件
将jsonl文件华为BIO格式
通过 BIO 格式数据构建知识图谱和本体,需要将实体和关系结构化存储,并定义概念、关系、以及图谱的逻辑。以下是完整的流程和实现方式:
从 BIO 数据中提取实体及其标签,组织成三元组
(subject, predicate, object) 格式。
定义领域内的概念(实体类型)、属性、关系规则等。
将提取的三元组存储在图数据库中(如 Neo4j),便于查询和可视化。
通过图数据库查询知识图谱,验证结果并进行推理。
从目前来看,有事件,时间,地点等信息了,但是目前来看人工标注太繁琐,因此需要将自动标注和人工标注合在一起使用
流程如下:
1.自动标注 → 2. 生成初始 BIO 数据 → 3. 人工修正 → 4. 实体与关系提取 → 5. 知识图谱构建与应用
工具和方法推荐
工具:
自动标注:Hugging Face Transformers、HanLP、spaCy。
手动补充:Doccano 或直接修改 BIO 数据。
存储:
轻量存储:使用 Pandas 或 NetworkX。
复杂场景:使用 RDF 或 Neo4j。